Krankheiten: Epidemieausbreitung auf einen Blick
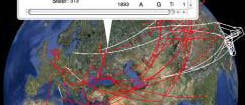
© Google Earth (Ausschnitt)
Eine globale Online-Landkarte könnte Wissenschaftlern bald helfen, Ausbrüche infektiöser Krankheiten genauer vorherzusagen und zu verfolgen. Die so genannte Supramap, die am 9. April online in der Fachzeitschrift Cladistics vorgestellt wurde, visualisiert die Ausbreitung von Krankheitserregern und ihrer Mutationen in Raum und Zeit ebenso wie in verschiedenen Wirtsorganismen. Bei der Realisierung des Projekts bedienten sich Forscher der Ohio State University (OSU) in Columbus und des American Museum of Natural History in New York unter anderem der Google Earth-Software. Diese berechnet aus Satellitendaten und Luftbildern einen virtuellen Globus und stellt ihn online dar.
"Im wesentlichen ähnelt das Ergebnis einer Wetterkarte, nur dass sie von Krankheiten handelt", sagt Daniel Janies, Professor für biomedizinische Informatik an der OSU, in einem eigens für die Vorstellung der Supramap produzierten youtube-Video. "Und Google Earth hilft uns, die Daten geeignet darzustellen."
Supramap, vorgestellt von seinen Entwicklern
Janies hofft, eines Tages kontinuierlich genomische und zugehörige geografische Daten aus aller Welt zu erhalten, die dann über Nacht analysiert und schon am folgenden Morgen auf der Landkarte dargestellt werden. Vor allem auch Gesundheitsbehörden und Politiker könnten sie im Fall von Epidemien nutzen, um geeignete Eindämmungsstrategien zu entwickeln.
Kernbestandteil des Systems, das auf einem Hochleistungsrechner an der OSU und dem Ohio Supercomputer Center läuft, sind komplexe Algorithmen zur phylogenetischen Rekonstruktion. Sie erlauben es, die genetische Entwicklung eines sich durch Mutationen verändernden Organismus zu verfolgen. Füttert man das System mit Gensequenzen neu entdeckter Viren, integriert es diese in den Stammbaum der Erregerstämme und projiziert die Daten schließlich auf den Google-Earth-Globus. Zusammen mit Angaben über Zeitpunkt und Ort der Funde stellt die Supramap unter anderem die Ausbreitungsrouten unterschiedlicher Erregerstämme dar und blendet an Orten, wo Mutationen stattfanden, zusätzliche Informationen ein.
Seine Nützlichkeit muss das System noch beweisen
Nachdem die Anwendung ursprünglich für die Darstellung der Ausbreitung des Grippeerregers H5N1 entwickelt wurde, arbeiten die Forscher nun an der Aufbereitung von Daten zur Virusvariante H1N1. Je mehr davon sie einfüttern, desto detailliertere Analysen wird das System erlauben. So sollte es zum Beispiel möglich sein, das erstmalige Überspringen eines Virus vom Tier zum Menschen zeitlich und räumlich genauer einzukreisen. Und lässt sich die Ausbreitung eines Pathogens etwa mit Mustern des Vogelflugs in Verbindung bringen, könnte dies die Forscher in die Lage versetzen, den Ort des nächsten Krankheitsausbruchs mit größerer Sicherheit zu bestimmen.
Seine Nützlichkeit muss das System nun zwar erst noch beweisen. Das Grundprinzip aber, genomische mit geografischen Daten zu verknüpfen und auf vergleichsweise einfache und anschauliche Art darzustellen, hat in jedem Fall Zukunft. Das sehen auch die Entwickler der Supramap so – und träumen davon, dass sie eines Tages sogar Informationen über die Entwicklung der Biodiversität in der Erdvergangenheit oder die globale Evolution von Lebensformen darstellen wird.
"Im wesentlichen ähnelt das Ergebnis einer Wetterkarte, nur dass sie von Krankheiten handelt", sagt Daniel Janies, Professor für biomedizinische Informatik an der OSU, in einem eigens für die Vorstellung der Supramap produzierten youtube-Video. "Und Google Earth hilft uns, die Daten geeignet darzustellen."
Supramap, vorgestellt von seinen Entwicklern
Janies hofft, eines Tages kontinuierlich genomische und zugehörige geografische Daten aus aller Welt zu erhalten, die dann über Nacht analysiert und schon am folgenden Morgen auf der Landkarte dargestellt werden. Vor allem auch Gesundheitsbehörden und Politiker könnten sie im Fall von Epidemien nutzen, um geeignete Eindämmungsstrategien zu entwickeln.
Kernbestandteil des Systems, das auf einem Hochleistungsrechner an der OSU und dem Ohio Supercomputer Center läuft, sind komplexe Algorithmen zur phylogenetischen Rekonstruktion. Sie erlauben es, die genetische Entwicklung eines sich durch Mutationen verändernden Organismus zu verfolgen. Füttert man das System mit Gensequenzen neu entdeckter Viren, integriert es diese in den Stammbaum der Erregerstämme und projiziert die Daten schließlich auf den Google-Earth-Globus. Zusammen mit Angaben über Zeitpunkt und Ort der Funde stellt die Supramap unter anderem die Ausbreitungsrouten unterschiedlicher Erregerstämme dar und blendet an Orten, wo Mutationen stattfanden, zusätzliche Informationen ein.
Seine Nützlichkeit muss das System noch beweisen
Nachdem die Anwendung ursprünglich für die Darstellung der Ausbreitung des Grippeerregers H5N1 entwickelt wurde, arbeiten die Forscher nun an der Aufbereitung von Daten zur Virusvariante H1N1. Je mehr davon sie einfüttern, desto detailliertere Analysen wird das System erlauben. So sollte es zum Beispiel möglich sein, das erstmalige Überspringen eines Virus vom Tier zum Menschen zeitlich und räumlich genauer einzukreisen. Und lässt sich die Ausbreitung eines Pathogens etwa mit Mustern des Vogelflugs in Verbindung bringen, könnte dies die Forscher in die Lage versetzen, den Ort des nächsten Krankheitsausbruchs mit größerer Sicherheit zu bestimmen.
Seine Nützlichkeit muss das System nun zwar erst noch beweisen. Das Grundprinzip aber, genomische mit geografischen Daten zu verknüpfen und auf vergleichsweise einfache und anschauliche Art darzustellen, hat in jedem Fall Zukunft. Das sehen auch die Entwickler der Supramap so – und träumen davon, dass sie eines Tages sogar Informationen über die Entwicklung der Biodiversität in der Erdvergangenheit oder die globale Evolution von Lebensformen darstellen wird.

Schreiben Sie uns!
Beitrag schreiben