Lexikon der Biochemie: kombinatorische Synthesen
kombinatorische Synthesen, kombinatorische chemische und biochemische Verfahren zur Erzeugung molekularer Vielfalt für den Aufbau von Verbindungsbibliotheken. Solche Bibliotheken dienen einer innovativen Leitstruktursuche und -optimierung für Pharmaka, Komplexbildner, Supraleiter, Farbstoffe, neue Katalysatoren u.a. Die k. S. haben sich in der Peptidchemie entwickelt (multiple Peptidsynthese). Bei der Suche nach Wirkstoffen für die Entwicklung neuer Pharmaka interessiert nicht mehr nur die Struktur einer Substanz: eine durch rationelles Wirkstoffdesign aus der Leitstruktur entwickelte Verbindung kann nicht besser sein als der gegenwärtige Erkenntnisstand über diese Struktur. Der aktuelle empirische Ansatz bei der Suche nach neuen Verbindungen mit gewünschten Eigenschaften versucht vielmehr den natürlichen Selektionsprozess zu kopieren. Dafür ist es erforderlich, Tausende potenzieller Substanzen mit nichtrepetitiven Molekülresten gleichzeitig zu erzeugen. Während die traditionelle organische Chemie eine Verbindung nach der anderen synthetisiert, gelingt es mit den k. S. viele Substanzen mit definierter Struktur gleichzeitig herzustellen. Dabei steht nicht mehr eine definierte Zielstruktur im Blickfeld, sondern eine Gruppe von Verbindungen, d.h. es werden anstelle der Bildung eines Produktes AB aus zwei Edukten A und B strukturchemisch differierende Edukte vom Typ A (A1-Ax)mit solchem vom Typ B (B1-Bx) nach den Prinzipien der Kombinatorik umgesetzt, wobei jeder Baustein vom Typ A mit jedem vom Typ B reagiert. Generell unterscheidet man zwischen parallelen und kombinatorischen Ansätzen, wobei Einzelverbindungen oder definierte Mischungen unterschiedlicher Komplexität resultieren. Die technische Durchführung erfolgt sowohl parallel, in separaten Behältnissen bzw. Kompartimenten, als auch simultan in einer Mischung. Ist z.B. x = 10, so resultieren 100 Produkte, während bei entsprechender Erweiterung auf mehrstufige Synthesen, bei denen in jeder Reaktion alle möglichen Kombinationen erzeugt werden, eine große Palette von Produkten entsteht, die man alsProduktmatrixoder Produktbibliothek bezeichnet. Eine dreistufige Synthese mit je x = 10 Edukten der Typen A, B, C und D liefert schon 10.000 Produkte. Die Bibliotheksgröße wird in mehrstufigen k. S. linear durch die Zahl der eingesetzten Edukte und exponenziell durch die Zahl der Reaktionsschritte determiniert.
Bei der natürlichen Selektion wird aus einer evolutiv entstandenen Vielfalt ähnlicher Strukturen ausgewählt. Die experimentelle Suche nach einem Wirkstoff beginnt in der Regel mit einer Leitstruktur, die mit Hilfe durchsatzstarker in-vitro-Primärtests durchgeführt wird. In der Medizinischen Chemie werden solchen Testsystemen (Assays) dem jeweiligen Krankheitsbild zugrunde liegende Schlüsselmechanismen zugeordnet, wofür als molekulare Sonden Rezeptoren oder Enzyme dienen. Ohne Kenntnis der Identität der individuellen Substanzen einer Bibliothek kann durch den Einsatz eines mechanismusorientierten Hochdurchsatz-Screenings, HTS (abgeleitet von engl. high-throughput-screening), das auf spezifischen Rezeptoren, Ionenkanälen oder Enzymen basiert, die im Substanzpool enthaltene aktivste Verbindung identifiziert werden. Entsprechend automatisierte Testsysteme ermöglichen das Screening von Tausenden von Substanzen pro Tag, wobei sogar mit sehr geringen Substanzmengen (sowohl löslicher als auch trägergebundener Verbindungen) die in-vitro-Aktivität zuverlässig bestimmt werden kann. Die Trefferquote liegt unter 1%. Die resultierenden Primärbefunde (Treffer, Hits) werden einer chemischen und biologischen Evaluierung unterworfen, die die Anzahl positiver Kandidaten weiter drastisch reduziert. Die so ausgewählten Verbindungen werden dann der Leitstrukturoptimierungzugeführt. Nicht die Quantität, sondern vielmehr die strukturelle Diversität (Verschiedenartigkeit) und Qualität stehen im Blickpunkt der weiteren Optimierung. Im Idealfall sollte eine möglichst effiziente Stammverbindung zur Steigerung der Wirkspezifität und Wirkhöhe systematisch modifiziert werden, bis Kandidaten für toxikologische und klinische Untersuchungen schließlich zu einem Arzneistoff führen. Die Medizinische Chemie benötigt dafür in der Regel acht- bis zehntausend synthetische Verbindungen. Die k. S. bilden eine pragmatische Alternative zu der parallelen Generierung einer großen Zahl von Molekülen unterschiedlicher Strukturen mit weniger Synthesestufen. Will man z.B. drei Aminosäuren zu den möglichen 27 Tripeptiden kombinieren, so lässt sich die k. S. dieser 27 Zielprodukte an einem polymeren Träger auf neun separate Schritte reduzieren, wobei sich die drei wiederholenden Arbeitsgänge (Mischen, Synthetisieren und Neuportionieren) leicht automatisieren lassen (Abb. 1). Im Gegensatz dazu werden für die lineare Synthese 81 Synthesestufen benötigt.
Bei Bibliotheken, die aus Parallelsynthesen resultieren, ist das Auffinden der Struktur aktiver Verbindungen relativ leicht, da der Syntheseort mit der aufzubauenden Struktur verknüpft ist. Dies erreicht man z.B. durch Kompartimentierung (jedes eingesetzte Polymerkügelchen enthält eine diskrete Molekülart) oder man legt ein Matrizenfeld an. Beispiele hierfür sind photolithographische Synthesen auf funktionalisierten Glasoberflächen, bei denen eine Kompartimentierung durch einfaches Abdecken von Teilen der Platte möglich ist, oder die Synthese von Benzodiazepinderivaten an einer Polyethylen-Pinmatrix. Multiple parallele Synthesen können beispielsweise auch auf Mikrotiterplatten durchgeführt werden.
Die Strukturbestimmung aktiver, trägergebundener Substanzen kann massenspektrometrisch erfolgen. Für eine nachträgliche Identifizierung wurden auch verschiedene Reportersysteme beschrieben, die eine Strukturaufklärung über Codemoleküle ermöglichen. Damit ist es möglich, die chemischen Syntheseschritte auf jeder Stufe zurückzuverfolgen, wodurch deduktiv auf die Struktur der Treffer geschlossen werden kann. Codierungssysteme sind beispielsweise oligomere Peptid- oder Nucleotidsequenzen, die parallel zum Aufbau der Substanzbibliothek synthetisiert werden. Die Code-Identifizierung erfolgt durch Sequenzierung, Nucleotidsequenzen können vorher gegebenfalls durch PCR (Polymerasekettenreaktion) amplifiziert werden. Für einen binären, nichtsequenziellen Code können auch Halogenaromaten eingesetzt werden, die man jeweils vor der Anküpfung der einzelnen Synthesebausteine in geringen Konzentrationen mit dem Träger verknüpft, und zur Codeentzifferung oxidativ abspaltet, silyliert und gaschromatographisch bestimmt. Man kann aber auch eine Matrixcodierung mit Fluorophoren vornehmen, die eine Sortierung mit einem Zellsortierer ermöglichen. Bei nichtchemischen Codeserfolgt die Aufzeichnung der entsprechenden Syntheseschritte beispielsweise in einem Speicherchip, der durch Hochfrequenz-Signale beschrieben und dechiffriert werden kann.
Nachteilig bei der Anwendung k. S. für den Aufbau von Peptid- und Polynucleotidbibliotheken ist , dass Peptide leicht einem proteolytischen Abbau unterliegen, geringe Bioverfügbarkeit besitzen und aufgrund der repetitiven Monomereinheiten nur eine eingeschränkte Diversität erlauben. Darüber hinaus ist die Übersetzung einer Peptidleitstruktur in ein nichtpeptidartiges Pharmakon nicht einfach zu realisieren. Aus diesem Grund werden inzwischen nichtrepetitive niedermolekulare organische Substanzbibliotheken bevorzugt. Die Abwandlung der Peptid- oder Nucleinsäuregrundstruktur führte beispielsweise zu Peptoiden, Peptidnucleinsäuren, Oligocarbamaten und Oligopyrrolinonen.
Für die praktische Durchführung der k. S. werden heute flexibel einsetzbare, zentral automatisierte probenorientierte Mehrkomponentensysteme verwendet, die typischerweise aus einem zentralen Steuerrechner, kombiniert mit verschiedenen spezialisierten Peripheriegeräten, die von einem zentralen Roboterarm mit Proben versorgt werden, bestehen (Abb. 2). Entscheidend ist weiterhin die Technologie zur Aufreinigung und Charakterisierung der dargestellten Einzelverbindungen. Nicht eindeutig charakterisierte Verbindungen führen zur Häufung falsch positiv deklarierter Treffer im biologischen Primärscreening, die dann in die aufwendigen Abläufe des Sekundärscreenings und der Leitstrukturoptimierung gelangen.
Die Entscheidung für das Format einer Substanzbibliothek als Einzelverbindungen oder Substanzmischungen (Abb. 3) ist sowohl für die Synthese als auch für das biologische Screening von Bedeutung. Mit Mischungen lassen sich in kürzerer Zeit mehr Verbindungen synthetisieren und testen, da in solchen Mischungen alle Substanzen aber in annähernd äquimolaren Mengen vorliegen, müssen sie anschließend in einem zeitaufwendigen, "Dekonvolution" (Entfaltung) genannten Verfahren getrennt werden. Hierzu sind die wiederholte Resynthese und Testung von Subbibliotheken mit geringerer Komplexität erforderlich, um die gesuchte Einzelverbindung einzukreisen und schließlich zu identifizieren. Bibliotheken mit mehr als 10.000 Verbindungen lassen sich effizient nur als Mischungen herstellen, während Bibliotheken von Einzelsubstanzen in der Regel weniger als 1000 Verbindungen enthalten.
In der Peptid- und Oligonucleotidchemie dominiert eindeutig die Festphasensynthese, für alle niedermolekularen organischen Reaktionen sind k. S. in Lösung nutzbar. Im letztgenannten Fall ist die Automatisierung der Aufarbeitungs- und Reinigungsschritte deutlich schwieriger, die Methodik ist hier noch nicht im gleichen Grad ausgereift.
Zusätzlich zur Nutzung für die Wirkstoffsuche und Pharmakaentwicklung wird versucht, mit Hilfe der kombinatorischen Biosynthesen den natürlichen Selektionsprozess zu kopieren. Durch zufällige Mutation, Rekombination und Duplikation von "Biosynthesegenen" verschiedener Eltern-Organismen lassen sich prinzipiell "nichtnatürliche Naturstoffe" herstellen, wofür auch der horizontale Gentransfer zwischen verschiedenen Organismen genutzt werden kann. Als Beispiel sollen die Gene der Enzyme genannt werden, die für die Kondensation, Zyklisierung und weiterführende Reaktionen des Biosyntheseweges der Polyketide verantwortlich sind. Diese Gene sind in Genkassetten angeordnet, die einer Neukombination unterzogen werden können. Selbstverständlich führt nicht jede Kombination zu einem neuen Produkt, da die Aktivität und Stabilität der veränderten Enzyme nicht vorhergesehen und gewährleistet werden kann. Trotz solcher Einschränkungen wurden mehr als 25 neuartige Polyketide auf dem Wege der kombinatorischen Biosynthese gewonnen. Der Aufwand für die Vergrößerung des Naturstoffpools auf diesem Wege ist jedoch noch relativ groß. (in-vitro-Selektion, Aptamere)
Die k. S. haben sich in den vergangenen Jahren zu einem wesentlichen Bestandteil neuer strategischer Ansätze zur Identifizierung neuer Materialien oder Katalysatoren, aber insbesondere zur Herstellung therapeutisch interessanter Pharmaka entwickelt. Für eine weitere Steigerung der Effizienz ist die Einbeziehung moderner Informationsverarbeitung, Miniaturisierung, Automatisierung, Mikrofluidik und moderner Trenn-, Analysen- und Detektionsmethoden, Bioassayoptimierung und insbesondere die Einbeziehung verbesserter Methoden der kombinatorischen Biologie und Biochemie ein absolutes Erfordernis. [G. Jung (Hrsg.) Combinatorial peptide and nonpeptide libraries – A handbook for the search of lead structures. Verlag Chemie, Weinheim, 1996; S. Cheng et al., Bioorgan. Med. Chem. 4 (1996) 727-737; M. Eigen u. R. Rigler, Proc. Natl. Acad. Sci. USA91 (1994) 5.740-5.747; C. Khosla et al., Biotechnol. Bioeng. 52 (1996) 122-128; K.T. O'Neil u. R.H. Hoess, Curr. Op. Struct. Biol. 5 (1995) 443-449]
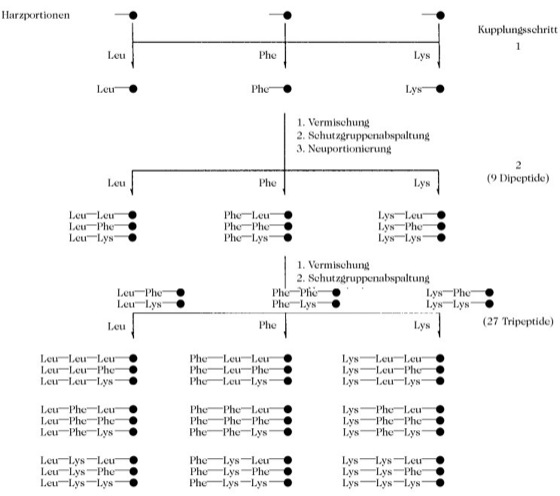
Kombinatorische Synthesen. Abb.1. Schematische Darstellung der Portionierungs-Mischungsmethode (engl. split and combine) am Beispiel einer kombinatorischen Tripeptidsynthese.

Kombinatorische Synthesen. Abb. 2. Schematische Vernetzung von Funktionalität in der probenorientierten Automation bei kombinatorischen chemischen Synthesen. A = Analytik (MS, HPLC, NMR etc.); HTS = Abk. für Hochdurchsatzscreening (engl. high throughput screening).

Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.