Lexikon der Biologie: Protein-Spleißen
Protein-Spleißen, protein splicing, autokatalytische Exzision eines internen Polypeptidsegments und Re-Ligation der flankierenden Enden zur Bildung eines funktionellen Proteins ( ß vgl. Abb. ). Das Herausschneiden des Peptidstücks wird in besonderen Enzymreaktionen von dem Protein selbst besorgt. Protein-Spleißen ist nach RNA-Spleißen (spleißen), RNA-trans-Spleißen, RNA-Editing und Recodierung eine weitere wichtige Entdeckung auf dem Gebiet der Genorganisation und Genexpression. Eine Vielzahl von Proteinen mit Spleißmechanismus ist inzwischen bekannt. Die meisten wurden bei Bakterien und primitiven Eukaryoten, wie z.B. der Backhefe, gefunden. In Vertebratenzellen kommt es bei der Prozessierung einiger Proteine auch zum Selbstspleißen (autosplicing), der anschließende Ligationsschritt bleibt jedoch aus. Bei den zuerst entdeckten derartigen Fällen, zum einen beim Hefe-Gen VMA/TFP1, das für eine vakuoläre ATPase codiert, sowie bei dem für die DNA-Polymerase codierenden Gen aus Thermococcus litoralis und dem recA-Gen aus Mycobacterium tuberculosis (Mykobakterien), wurde zunächst eine Insertion in hoch konservierten Genabschnitten gefunden, die eine deutliche Abweichung zu allen anderen bisher bekannten Genen der jeweiligen Familie darstellten. Die intervenierenden Sequenzen wurden zunächst als mögliche Intronen (Intron) eingestuft, jedoch erhalten sie überraschenderweise zwischen den flankierenden konservierten Aminosäuresequenzen über eine Strecke von mehreren hundert Codonen das Lese-Raster. Auf Proteinebene wurden in allen Fällen 2 Proteinprodukte der jeweiligen offenen Leseraster (open reading frame = ORF) nachgewiesen, die addiert die Molekülmasse des aus dem ORF vorherberechneten Einzelprodukts ergaben. Bei weiterer Analyse der beiden Proteine zeigte sich, daß eines einen internen Bereich des Proteins darstellte, während das andere die beiden außenliegenden Bereiche in sich vereinte (Inteine). In keinem der Fälle konnte RNA-Prozessierung nachgewiesen werden. Einführung von Lese-Rastermutationen im mittleren Bereich führte zum Verschwinden beider Proteine, und Veränderungen an dritten Codon-Positionen, welche die Aminosäuresequenz erhalten, auf Nucleinsäureebene jedoch eine starke Sequenzabweichung erzeugen, ließen die Proteinprodukte intakt. Im Gegensatz dazu führt der Austausch eines Codons an der vermuteten Grenze zwischen Protein-Intron und normaler Gensequenz zur Produktion eines Polypeptids mit der Molekülmasse, die der aus dem gesamten ORF vorhergesagten entspricht. Dies zeigt deutlich, daß die Aminosäure- und nicht die korrespondierende RNA-Sequenz für diese Form von Spleißen entscheidend ist. Da die Protein-Spleißen-Reaktion auch in vitro im heterologen System sowie in vivo bei Expression im heterologen E.-coli-System funktionell bleibt, wird ein autokatalytischer Mechanismus angenommen. Auch scheint das Protein-Intron allein ausreichend für die katalytische Reaktion, da auch nach Entfernen fast der ganzen umgebenden „Protein-Exon“-Sequenz das Spleißen intakt bleibt. – Neben der Protein-Spleißen-Aktivität des „spacer“-Proteins enthält dieses noch eine hochspezifische Endonuclease-Aktivität mit Homologie zu H0-Endonuclease aus dem Mating-type-System von Hefe. Ebenfalls homolog dazu sind Endonucleasen, die einzeln sowie in Intronen der Gruppe I vorgefunden werden. Endonucleasen, die in Intronen der Gruppe I codiert werden, erkennen eine Sequenz auf der DNA, die in der Nähe der Intron-Insertionsstelle liegt. Wenn nun das Intron im Gen insertiert ist, wird diese Erkennungsstelle unterbrochen und läßt damit die DNA für die Endonuclease unzugänglich. Reparatur durch die von der Intron-codierten Endonuclease geschnittene DNA ohne Intron mit homologer, nicht geschnittener, weil Intron enthaltender DNA als Matrize führt nun zu einem unidirektionalen Gentransfer von einem Gen mit Intron in ein „leeres“ Gen ohne Intron. Dieses Phänomen wird Intron-homing genannt und führt sowohl zur Erhaltung von Intronen der Gruppe I an ihrer Position in entsprechenden Genen als auch zu deren Verbreitung an neue Stellen. Auch bei den Protein-Intronen wurde gezeigt, daß die „spacer“-Proteine mit ihrer Endonuclease-Aktivität (homing-Endonuclease) die entsprechende DNA-Sequenz, die das korrespondierende Gen ohne Insertion auf DNA-Ebene enthält, spalten. Darüber hinaus konnte auch hier ein Homing der Protein-Intron codierenden DNA-Sequenz in die Schnittstelle (über DNA-Rekombinationsreparatur) in vivo gezeigt werden. Das Auftreten in allen drei phylogenetischen Grundunterteilungen läßt eine sehr breite Verteilung des Protein-Spleißens vermuten. Über den Ursprung von Protein-Spleißen können bisher nur Vermutungen angestellt werden, wobei die beiden extremsten Hypothese-Varianten zum einen einen sehr ursprünglichen Mechanismus (deshalb in allen Organismenreichen vertreten) und zum anderen einen sehr modernen Mechanismus (die weite Verbreitung findet über horizontalen Gentransfer statt) postulieren. Obwohl bis jetzt noch nicht gezeigt, ist nicht auszuschließen, daß einige der Protein-Intronen ihre Protein-Spleißen-Aktivität verloren haben und abhängig von anderen nun in trans agierenden „spacer“-Proteinen sind. Solche Ziel-Intronen könnten viel ihrer internen Sequenz verlieren und müßten lediglich die essentiellen Erkennungsstellen erhalten. Solche degenerierten Protein-Intronen könnten dann sehr klein sein und auch, nach Ausschneiden, nur zu marginalen Unterschieden in der Molekülmasse und Aminosäuresequenz des von der DNA-Sequenz vorhergesagten Proteins führen. Mögliche „Kandidaten“ hierfür sind schon in Untersuchung. – Bei dem als Hedgehog (hedgehog) bezeichneten Protein der Taufliege Drosophila melanogaster, einem Protein der Steuerung der Embryonalentwicklung, wurden Elemente des Protein-Spleißens nachgewiesen. Zunächst entsteht eine intakte Vorstufe des Proteins. In einer sich selbst katalysierenden Reaktion wird dann am hinteren Ende des Moleküls ein Stück abgespalten und gleichzeitig an das neu entstandene Ende der vorderen Molekülhälfte ein Cholesterinmokekül angehängt, mit dem das Molekül in der Zellmembran verankert wird. Die an der Umgestaltung des Hedgehog-Proteins beteiligten Katalysatoren sind – ähnlich wie bei den Inteinen – Teil des umzuformenden Moleküls.
P.Z./S.Kl.
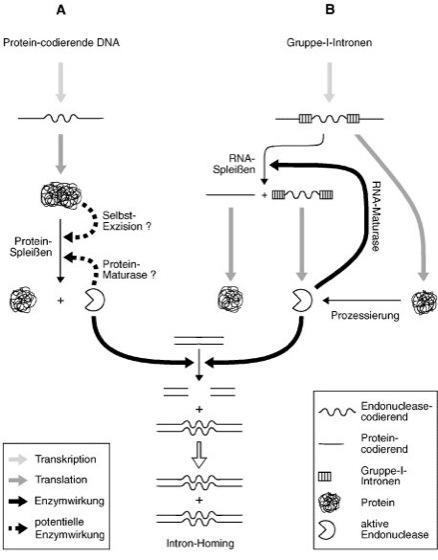
Protein-Spleißen
A Protein-Spleißen: translatiertes Protein spleißt sich selbst oder wird durch Maturase (in trans) gespleißt. B Gruppe-I-Intronen: translatiertes Exon-Intron-Proteinprodukt wird über proteolytische Prozessierung in Maturase überführt, die zum RNA-Spleißen benötigt wird. A und B: sowohl „spacer“-Protein als auch Maturase zeigen Endonuclease-Aktivität, die Intron-homing bewirkt.
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.