Molekulardynamik: Die Geheimnisse des Lebens berechnen
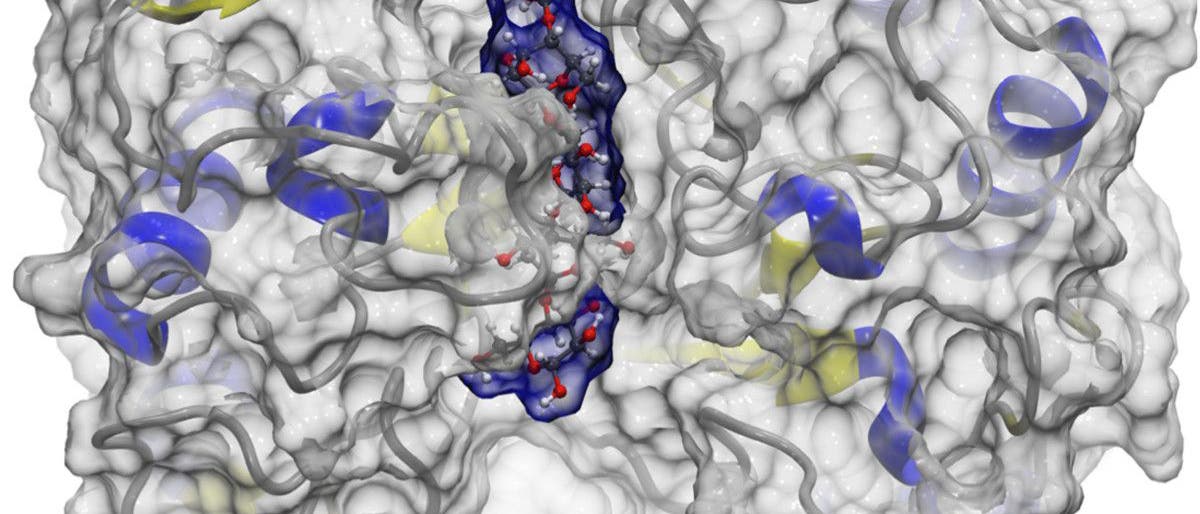
In den letzten 40 Jahren haben die Computer einen Faktor 109 (eine Milliarde) an Leistungsfähigkeit zugelegt. Damit sind sie zwar in der Lage, immer aufwändigere Berechnungen anzustellen; aber ist die Welt so komplex, dass man diese großen und teuren Geräte wirklich braucht? Was die Lebenswissenschaften angeht, so können wir diese Frage mit einem klaren Ja beantworten. Wesentliche Prozesse finden auf der Ebene der Moleküle statt; die entscheidenden Akteure innerhalb einer Zelle sind die Proteine. Sie katalysieren chemische Reaktionen aller Art, zum Beispiel beim Verarbeiten der Nahrung, wandeln bei der Fotosynthese Licht in chemische Energie um, geben als Stützstrukturen einzelnen Zellen und ganzen Körpern Halt und erledigen noch viele andere Aufgaben. Unser Erbgut besteht zu einem wesentlichen Teil aus Bauanleitungen für Proteine.
In der Rohform bestehen sie aus Ketten von einigen hundert Aminosäuren mit insgesamt mehreren tausend Atomen. Aber ihre Wirkungen entfalten sie erst, indem sie eine spezielle räumliche Struktur annehmen. Obendrein pflegen sie bei der Arbeit ihre Form zu ändern, und in aller Regel arbeiten mehrere Proteine in engem Kontakt an einer Aufgabe zusammen, gleichsam als molekulare Maschinen mit mehreren Bestandteilen. Schließlich spielt die Umgebung wie zum Beispiel die wässrige Lösung, in der sich die Reaktionen abspielen, eine wesentliche Rolle – und schon sind nicht nur einige tausend, sondern Millionen Atome beteiligt.
Die genaue Funktionsweise dieser Maschinen lässt sich mit einer einzelnen Methodik praktisch nicht aufklären. Wer die dynamischen Prozesse im Reagenzglas verfolgt, kann herausfinden, wie sich die Konzentration der Reaktionsteilnehmer mit der Zeit verändert. Wer misst, wie die Atome eines Proteins gegeneinander schwingen, kann aus der Veränderung des Schwingungsmusters erschließen, welche Bereiche des Moleküls sich verändern. Man kann es kristallisieren – was nur bei kleinen Proteinen gelingt – und das Beugungsmuster der Röntgenstrahlen analysieren; man kann es einfrieren und mit dem Elektronenmikroskop untersuchen. Die beiden zuletzt genannten experimentellen Verfahren liefern im günstigen Fall ein sehr genaues Bild von der räumlichen Gestalt ("Konformation") des Moleküls, aber eben nur eine Momentaufnahme. Man kann also gewissermaßen studieren, was die molekulare Maschine herstellt und wie sie zu speziellen Zeitpunkten aussieht. Daraus zu erschließen, wie sie funktioniert, ist schon bei einer gewöhnlichen Maschine eine Herausforderung.
Genau das ist uns nun bei etlichen molekularen Maschinen gelungen – mit Hilfe der Computersimulation. ...
Schreiben Sie uns!
Beitrag schreiben