Zellbiologie: Gläsernes Bakterium
© Takuji Yamada, SCB, EMBL (Ausschnitt)
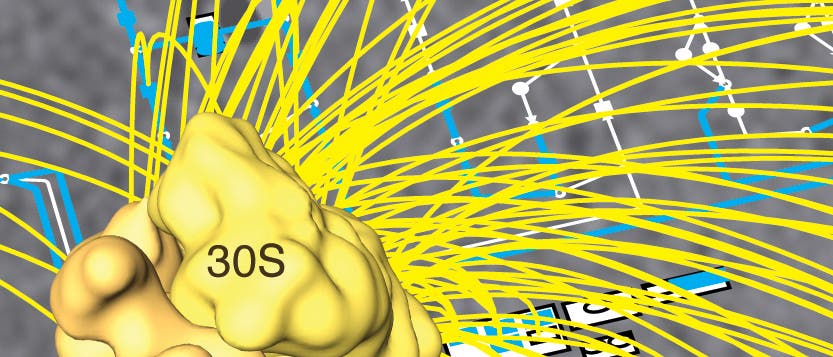
Einem internationalen Forscherteam ist es jetzt gelungen, praktisch sämtliche Zellvorgänge innerhalb des Bakteriums Mycoplasma pneumoniae zu katalogisieren. Die Wissenschaftler erfassten dazu die Gesamtheit der Proteine, RNA-Abschriften und Stoffwechselvorgänge. Das Genom von M. pneumoniae – einem Erreger der atypischen Lungenentzündung – ist selbst für ein Bakterium winzig und zählt zu den kleinsten, die man kennt. Die Forscher erhoffen sich von ihren Untersuchungen deshalb auch Aufschluss über die Frage nach der Mindestausstattung eines lebensfähigen Organismus.
Geleitet wurde die Dreifachstudie von Peer Bork und Anne-Claude Gavin vom European Molecular Biology Laboratory in Heidelberg und ihrem ehemaligen Kollegen Luis Serrano, jetzt am Centre de Regulacio Genòmica in Barcelona. Eine Gruppe analysierte das Transkriptom, also sämtliche in der Zelle kursierenden RNA-Abschriften von Genen, unter verschiedenen Umweltbedingungen. Eine zweite erfasste das Metabolom – die Gesamtheit der Stoffwechselvorgänge innerhalb von M. pneumoniae – unter den gleichen Bedingungen. Die dritte Gruppe konzentrierte sich auf das so genannte Proteom und damit auf die Frage, welche Proteine wann gebildet werden und wie sie miteinander interagieren.
Wie die Forscher berichten, erwies sich das Innenleben des Bakteriums trotz der Kürze des DNA-Strangs als überraschend vielfältig: Zahlreiche Proteine würden beispielsweise eine Doppelfunktion übernehmen und sich mit verschiedenen Partnern zu Komplexen zusammenfinden. Auch bei der Genregulation habe der Mikroorganismus Wege gefunden, den Mangel an spezialisierten Steuerungsmolekülen wettzumachen. In vielerlei Hinsicht zeige das Bakterium eine Zellorganisation, die der von Zellen mit Zellkern (Eukaryoten) ähnele, urteilen Howard Ochman und Rahul Raghavan von der University of Arizona in einem Begleitkommentar. M. pneumoniae sei "randvoll mit hochkomplexen Regulationsnetzen und innovativen Signalwegen". All das lege den Schluss nahe, dass es "simple Bakterien" nicht gebe.
Jan Dönges
Geleitet wurde die Dreifachstudie von Peer Bork und Anne-Claude Gavin vom European Molecular Biology Laboratory in Heidelberg und ihrem ehemaligen Kollegen Luis Serrano, jetzt am Centre de Regulacio Genòmica in Barcelona. Eine Gruppe analysierte das Transkriptom, also sämtliche in der Zelle kursierenden RNA-Abschriften von Genen, unter verschiedenen Umweltbedingungen. Eine zweite erfasste das Metabolom – die Gesamtheit der Stoffwechselvorgänge innerhalb von M. pneumoniae – unter den gleichen Bedingungen. Die dritte Gruppe konzentrierte sich auf das so genannte Proteom und damit auf die Frage, welche Proteine wann gebildet werden und wie sie miteinander interagieren.
Wie die Forscher berichten, erwies sich das Innenleben des Bakteriums trotz der Kürze des DNA-Strangs als überraschend vielfältig: Zahlreiche Proteine würden beispielsweise eine Doppelfunktion übernehmen und sich mit verschiedenen Partnern zu Komplexen zusammenfinden. Auch bei der Genregulation habe der Mikroorganismus Wege gefunden, den Mangel an spezialisierten Steuerungsmolekülen wettzumachen. In vielerlei Hinsicht zeige das Bakterium eine Zellorganisation, die der von Zellen mit Zellkern (Eukaryoten) ähnele, urteilen Howard Ochman und Rahul Raghavan von der University of Arizona in einem Begleitkommentar. M. pneumoniae sei "randvoll mit hochkomplexen Regulationsnetzen und innovativen Signalwegen". All das lege den Schluss nahe, dass es "simple Bakterien" nicht gebe.
Jan Dönges

Schreiben Sie uns!
Beitrag schreiben