Nobelpreise 2014: Bilder von der Grenze zwischen Biologie und Chemie
Auch physikalische Gesetze sind manchmal etwas weniger absolut als sie scheinen. Als der Physiker Stefan Hell sich vornahm, die Beugungsgrenze zu überwinden, sahen die meisten Fachkollegen in der 1873 von Ernst Abbe formulierten Gesetzmäßigkeit eine unüberwindbare Hürde: Lichtmikroskope würden niemals Strukturen unterhalb von etwa 200 Nanometern Durchmesser auflösen, der halben Wellenlänge des sichtbaren Lichts. Das war, wie sich zeigte, ein Irrtum. Für den Nachweis, dass Lichtmikroskope Details weit unterhalb dieser Grenze darstellen können, erhielt Hell dieses Jahr zusammen mit Eric Betzig und W.E. Moerner den Nobelpreis für Chemie. Die Bilder, die ihre Methoden erzeugen, zeigen die Welt an der Grenze zwischen Chemie und Biologie.
Das Beugungslimit ist eine grundlegende Eigenschaft der Optik. Zwei Punkte, die weniger als eine halbe Lichtwellenlänge voneinander sind, zeigen sich als einzelner, verschmierter Punkt. Umgekehrt ist es auch nicht möglich, Licht auf einen Punkt mit weniger als der halben Wellenlänge Durchmesser zu fokussieren. Bei sichtbarem Licht sind das etwa 200 Nanometer – etwa die Größe einer Zellorganelle. Unterhalb dieser Grenze sind normale Lichtmikroskope blind.
Jenseits der Beugungsgrenze
Für die Biologie ist die Beugungsgrenze ein echtes Problem. Um zu verstehen, wie eine Zelle funktioniert, muss man einzelne Moleküle im Blick behalten, die zehn oder hundert Mal kleiner sind als diese magische Grenze. Zwar ist das Beugungslimit für das Elektronenmikroskop kein Problem, und andere Techniken wie die Röntgenbeugung, zeigen sogar einzelne Atome. Doch diese Verfahren benötigen harsche Bedingungen, zum Beispiel Hochvakuum und energiereiche Strahlen, die jedes Leben zuverlässig töten. Das Lichtmikroskop dagegen zeigt das Leben in voller Blüte, so lange man will. Nur eben nicht in hoher Auflösung.
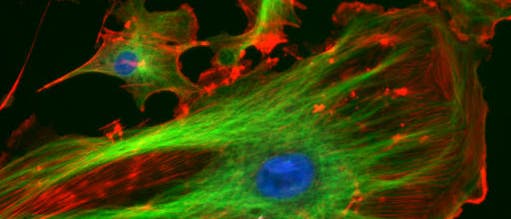
Sogar noch mehr Details zeigt das Lichtmikroskop in Zusammenarbeit mit fluoreszenzmarkierten Antikörpern. Mit deren Hilfe lokalisieren Forscher Biomoleküle innerhalb der Zelle, um ihre Funktion zu entschlüsseln. Das Prinzip ist einfach: Bestimmte Moleküle lassen sich mit einem Laser anregen, ihre Elektronen steigen in einen energiereicheren Zustand auf. Anschließend fallen sie wieder zurück und geben ein Photon ab – sie leuchten.
Koppelt man solche Moleküle an Antikörper, die bestimmte Biomoleküle binden, und behandelt eine Zelle damit, kann man sehen, wo diese Moleküle in der Zelle liegen – dass sich bestimmte Proteine nahe der Zellmembran anordnen zum Beispiel. Doch Details unterhalb des Beugungslimits blieben weiterhin verborgen.
Die Fluoreszenz lieferte Hell jedoch das Werkzeug, mit dem er das Beugungslimit aus den Angeln hob. Mit einem quantenmechanischen Trick sorgte er dafür, dass nur Moleküle an einem ganz bestimmten Punkt mit wenigen Nanometern Durchmesser Licht abgeben können. Der Anregungslaser, in dessen Strahl die markierten Proteine zu leuchten beginnen, kann zwar nicht unter das Beugungslimit fokussiert werden, doch Hell erkannte, dass unter bestimmten Bedingungen nur Proteine genau im Strahlzentrum Licht abgeben.
Ein quantenmechanischer Trick
Dafür verantwortlich ist die so genannte stimulated emission depletion (STED, stimulierte Emissionsleerung), die den Prozess der Fluoreszenz unterbindet. Bei der normalen Fluoreszenz hebt das Laserlicht ein Elektron in einen angeregten Zustand. Anschließend fällt dieses Elektron wieder in den Grundzustand zurück und gibt ein Photon ab. Doch ein zweiter Laser mit niedrigerer Energie zwingt die angeregten Elektronen auf einen anderen Weg – sie fallen in einen anderen Zustand zurück, der eine höhere Energie hat als der Grundzustand, so dass ihr Licht eine andere Farbe hat und herausgefiltert werden kann.
Bei der von Hell entwickelten STED-Mikroskopie erreicht man das mit einem zweiten Laser mit höherer Wellenlänge, der in den Außenbereichen des Lichtflecks vom Anregungslaser einen eigenen, ringförmigen Lichtfleck erzeugt. Dort leert das zusätzliche Licht die angeregten Zustände der markierten Moleküle quasi aus. Nur im Inneren des Rings, im Zentrum des Anregungslasers, können noch Moleküle fluoreszieren.
Mit dieser winzigen Sonde fährt man nun die Probe Nanometer für Nanometer ab – und erreicht so eine Auflösung weit jenseits der physikalischen Grenzen des Lichtmikroskops, einfach weil der Mechanismus nicht mehr von der Beugung abhängig ist. Zwei Punkte innerhalb des Beugungslimits erscheinen als getrennt, weil je einer von ihnen durch den zweiten Laser am Leuchten gehindert wird.
Wo Hell den Bereich einschränkte, aus dem er Signale empfing, gingen W.E. Moerner und Eric Betzig den anderen Weg – sie stellten sicher, dass sie wirklich nur das Licht einzelner Moleküle maßen. Sobald man weiß, dass ein Lichtpunkt aus exakt einem Molekül besteht, kann man dieses trotz Beugungslimit extrem genau lokalisieren. Vorausgesetzt, die Moleküle sind nicht näher als die halbe Lichtwellenlänge voneinander entfernt.
Doch in Zellen liegen die interessanten Biomoleküle viel, viel dichter gepackt – sie jemals einzeln zu erwischen, schien ausgeschlossen. Bis W.E. Moerner in den 1990er Jahren ein ganz besonderes Molekül entdeckte.
Es handelte sich damals um eine Variante des Green Fluorescent Protein, für das Roger Y. Tsien eine Dekade später den Nobelpreis erhielt. Dieses Protein, und die gesamte Klasse an Molekülen, die aus ihm hervorging, fluoresziert – doch Moerners Entdeckung tat noch mehr: Die Fluoreszenz ließ sich aus- und wieder anschalten. Mit Licht einer Wellenlänge von 488 Nanometern bestrahlt, leuchtet das Molekül. Aber nicht lange, dann erlischt es und lässt sich auch durch weitere Anregung mit diesem Licht nicht wieder erwecken. Wie Moerner aber feststellte, dreht ein Lichtpuls anderer Farbe – 405 Nanometer – die Uhr des Moleküls zurück, so dass es bei Anregung wieder für eine Weile aufscheint.
Schaltbare Farbstoffe
Damit ebnete Moerner den Weg zu Betzigs späterer Entwicklung. Dessen zentrale Erkenntnis: Wenn man oft nacheinander jeweils nur einen Bruchteil der Moleküle fluoreszieren lässt, sind die leuchtenden Proteine schon aus statistischen Gründen weit voneinander entfernt, und damit präzise lokalisierbar. Das neue, von Moerner gefundene Protein bot ihm die Möglichkeit, das theoretische Prinzip in die Praxis zu überführen.
Bei der Einzelmolekülmikroskopie bestrahlt man die Probe mit dem Anregungslicht, bis alle fluoreszierenden Moleküle erschöpft und dunkel sind. Die nun im Aus-Zustand befindlichen Markersubstanzen setzt man anschließend dem Anschaltpuls aus – allerdings nur mit sehr geringer Intensität. Das schwache Licht enthält schlicht nicht genug Photonen, um mehr als einen Bruchteil der Fluoreszenz-Moleküle wieder anzuschalten, so dass sie für eine kurze Weile leuchten. So sind die Leuchtpunkte im Mikroskop alle weiter voneinander entfernt als das Beugungslimit – entsprechend weiß man auf wenige Nanometer genau, wo die zugehörigen Biomoleküle sind.
Natürlich enthüllt ein einzelnes Bild keine Details der Struktur – dazu sieht man zu wenig der Moleküle. Doch wenn man den Prozess vielfach wiederholt, lässt sich im Laufe der Zeit aus den einzelnen Bildern ein Gesamtbild mit fast allen fluoreszenzmarkierten Molekülen zusammensetzen – weit höher aufgelöst, als es die Beugungsgrenze jemals erlauben würde.
Die Strukturen, die man mit den von den drei Nobelpreisträgern entwickelten Verfahren sieht, stammen aus jenem bisher unzugänglichen Bereich, in dem Biologie zu Chemie wird – auf dem Level des einzelnen Moleküls, doch eingebettet in jene Strukturen, in denen es seine Funktion erfüllt. Seit Jahren vermuten Forscher, dass die räumliche Anordnung verschiedener Proteine für viele komplexe Reaktionsketten des Lebens von entscheidender Bedeutung ist – für die Funktion von Synapsen im Gehirn ebenso wie für die Fotosynthese oder auch das Auslesen des Erbgutes mit Hilfe ganzer Proteinkomplexe. Die hochaufgelöste Fluoreszenzmikroskopie erlaubt nun einen direkten Blick in diese Maschinerie des Lebens.
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.