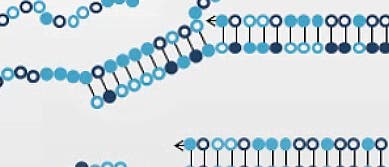
DNA-Computing: Forscher entwickeln Künstliches Neuronales Netz aus DNA-Strängen
© Lulu Qian (Ausschnitt)
Neue Möglichkeiten bei der Entwicklung kleiner Rechenmaschinen aus DNA öffnet die Forschungsarbeit eines Teams des California Institute of Technology in Pasadena: Es gelang den Forschern, ein so genanntes Künstliches Neuronales Netz aus DNA-Strängen zu bauen. Solche Netze, die grob der Arbeitsweise des Gehirns nachempfunden sind, haben gegenüber einer klassischen Computerarchitektur den Vorteil, auch unvollständige oder gestörte Eingaben verarbeiten zu können. Die Netze suchen dabei nach Ähnlichkeiten mit eingespeicherten Eingabemustern.
Das Team um Lulu Qian orientierte sich am einfachsten Modell eines solchen Netzwerks: Es besteht aus Knoten – den "Neuronen" –, die entweder den Wert null (inaktiv) oder eins (aktiv) annehmen können, und gewichteten Verbindungen zwischen den Knoten. Ob die Knoten aktiv oder inaktiv sind, richtet sich nach der Aktivität derjenigen Nachbarn, mit denen sie verknüpft sind. Die Verbindungsgewichte aller Verbindungen mit aktiven Einheiten werden dabei aufsummiert und mit einem Schwellenwert verglichen. Überschreitet die Summe die Schwelle, erhält das Neuron den Wert eins.
Zwei Knoten, die beispielsweise durch eine Verbindung mit stark positivem Gewicht verbunden sind, erregen sich dadurch gegenseitig und neigen dazu, am Ende gleichzeitig aktiv zu sein. Ist das Gewicht dagegen stark negativ, kann das eine der beiden Neurone das andere unterdrücken, also auf null setzen. Durch geschickte Wahl der Verbindungsgewichte und Schwellenwerte können auf diese Weise Muster gleichzeitig aktiver oder inaktiver Neurone in der Struktur des Netzwerks gespeichert werden. Aktiviert man nun als Eingabe einige der Neurone und lässt sie sich gegenseitig hemmen und erregen, pendelt sich mit der Zeit die Aktivierung in demjenigen eingespeicherten Muster ein, das dem der Eingabe am stärksten ähnelt.
Ein solches so genanntes Hopfield-Netz konstruierten nun Qian und Kollegen Erik Winfree und Jehoshua Bruck aus DNA, wenn auch nur mit lediglich vier Kunstneuronen. Sie griffen dabei auf eine selbst entwickelte Methode zurück, deren Nützlichkeit sie kürzlich durch den Bau klassischer UND- und ODER-Logikgatter unter Beweis stellten. Das Verfahren baut auf Elementen auf, die die Forscher als See-Saw-Gates bezeichnen.
See-Saw-Gates bestehen aus doppelsträngiger DNA, deren einer Strang von einem besser passenden "Eingabe"-Einzelstrang verdrängt werden kann und dadurch als "Ausgabe" das Gate verlässt. Verschieden hohe Konzentrationen der entsprechenden DNA-Abschnitte und Gates repräsentierten die Werte der Verbindungsgewichte und Schwellenwerte. Nach mehreren Stunden des miteinander Reagierens zeigten zusätzlich angebrachte Fluoreszenzmarker schließlich, zu welchem Ergebnis das Netz gekommen war.
Das Neuronale Netz des Forscherteams um Lulu Qian hat neben der sehr langen Reaktionsdauer den Nachteil, dass es nur einmal verwendbar ist. Danach müssen die 112 verschiedenen Strangtypen, die für das Vier-Neurone-Netz notwendig waren, neu synthetisiert werden. Überdies fehlt ihm die Lernfähigkeit, die normalerweise solche Neuronalen Netze auszeichnet. Welche Verbindungsgewichte und Schwellenwerte für die einzuspeichernden Muster geeignet waren, musste das Wissenschaftlerteam vorab durch Computersimulationen bestimmen.
Dennoch sind die Forscher zuversichtlich, mit ihrem Ansatz auf absehbare Zeit einen molekularen Einfachst-Computer entwickeln zu können, der sich im Unterschied zur klassischen Siliziumtechnologie auf natürliche Weise in biologische Systeme integrieren lassen sollte. Im Innern einer Zelle könnte er, so jedenfalls die Hoffnung der Wissenschaftler, das Vorhandensein oder die Konzentration verschiedener Signalmoleküle gleichzeitig ermitteln und programmgemäß darauf reagieren. Gerade bei einem solchen Einsatz dürften der DNA-Maschine die robuste Mustererkennung und Fehlertoleranz eines Künstlichen Neuronalen Netzes zu Gute kommen. (jd)
Das Team um Lulu Qian orientierte sich am einfachsten Modell eines solchen Netzwerks: Es besteht aus Knoten – den "Neuronen" –, die entweder den Wert null (inaktiv) oder eins (aktiv) annehmen können, und gewichteten Verbindungen zwischen den Knoten. Ob die Knoten aktiv oder inaktiv sind, richtet sich nach der Aktivität derjenigen Nachbarn, mit denen sie verknüpft sind. Die Verbindungsgewichte aller Verbindungen mit aktiven Einheiten werden dabei aufsummiert und mit einem Schwellenwert verglichen. Überschreitet die Summe die Schwelle, erhält das Neuron den Wert eins.
Zwei Knoten, die beispielsweise durch eine Verbindung mit stark positivem Gewicht verbunden sind, erregen sich dadurch gegenseitig und neigen dazu, am Ende gleichzeitig aktiv zu sein. Ist das Gewicht dagegen stark negativ, kann das eine der beiden Neurone das andere unterdrücken, also auf null setzen. Durch geschickte Wahl der Verbindungsgewichte und Schwellenwerte können auf diese Weise Muster gleichzeitig aktiver oder inaktiver Neurone in der Struktur des Netzwerks gespeichert werden. Aktiviert man nun als Eingabe einige der Neurone und lässt sie sich gegenseitig hemmen und erregen, pendelt sich mit der Zeit die Aktivierung in demjenigen eingespeicherten Muster ein, das dem der Eingabe am stärksten ähnelt.
Ein solches so genanntes Hopfield-Netz konstruierten nun Qian und Kollegen Erik Winfree und Jehoshua Bruck aus DNA, wenn auch nur mit lediglich vier Kunstneuronen. Sie griffen dabei auf eine selbst entwickelte Methode zurück, deren Nützlichkeit sie kürzlich durch den Bau klassischer UND- und ODER-Logikgatter unter Beweis stellten. Das Verfahren baut auf Elementen auf, die die Forscher als See-Saw-Gates bezeichnen.
See-Saw-Gates bestehen aus doppelsträngiger DNA, deren einer Strang von einem besser passenden "Eingabe"-Einzelstrang verdrängt werden kann und dadurch als "Ausgabe" das Gate verlässt. Verschieden hohe Konzentrationen der entsprechenden DNA-Abschnitte und Gates repräsentierten die Werte der Verbindungsgewichte und Schwellenwerte. Nach mehreren Stunden des miteinander Reagierens zeigten zusätzlich angebrachte Fluoreszenzmarker schließlich, zu welchem Ergebnis das Netz gekommen war.
Das Neuronale Netz des Forscherteams um Lulu Qian hat neben der sehr langen Reaktionsdauer den Nachteil, dass es nur einmal verwendbar ist. Danach müssen die 112 verschiedenen Strangtypen, die für das Vier-Neurone-Netz notwendig waren, neu synthetisiert werden. Überdies fehlt ihm die Lernfähigkeit, die normalerweise solche Neuronalen Netze auszeichnet. Welche Verbindungsgewichte und Schwellenwerte für die einzuspeichernden Muster geeignet waren, musste das Wissenschaftlerteam vorab durch Computersimulationen bestimmen.
Dennoch sind die Forscher zuversichtlich, mit ihrem Ansatz auf absehbare Zeit einen molekularen Einfachst-Computer entwickeln zu können, der sich im Unterschied zur klassischen Siliziumtechnologie auf natürliche Weise in biologische Systeme integrieren lassen sollte. Im Innern einer Zelle könnte er, so jedenfalls die Hoffnung der Wissenschaftler, das Vorhandensein oder die Konzentration verschiedener Signalmoleküle gleichzeitig ermitteln und programmgemäß darauf reagieren. Gerade bei einem solchen Einsatz dürften der DNA-Maschine die robuste Mustererkennung und Fehlertoleranz eines Künstlichen Neuronalen Netzes zu Gute kommen. (jd)



Schreiben Sie uns!
Beitrag schreiben