DNA-Origami: Nanomaschinen aus dem Steckbaukasten
Wer einmal aus einem Blatt Papier einen Kranich, einen Frosch oder einen Hut gefaltet hat, weiß: Origami, die Kunst des Papierfaltens, ist so einfach wie genial. Mit nur einigen wenigen klar definierten Falt- und Knickfolgen lassen sich unfassbar filigrane und raffinierte dreidimensionale Formen erstellen. Was aber, wenn nun statt Papier Stränge aus DNA gefaltet werden? Und das Ergebnis keine hübsch anzusehenden, weitgehend nutzlosen Figürchen sind, sondern winzige funktionale Strukturen, die als Nanomotoren, als künstliche Zellstrukturen oder gar als antivirale Medikamente eingesetzt werden können?
Der Mann, der daran arbeitet, dass all das schon bald Wirklichkeit wird, sitzt in seinem Büro auf dem Garchinger Campus am Stadtrand von München. Begeistert zeigt er auf die Videoanimation eines großen Haufens verworrener Moleküle. Normalerweise ist Hendrik Dietz kein Mann für verschlungene Pfade und Schnickschnack. Seine rasante Forscherkarriere verlief schnurgerade. Physikdiplom 2004, Promotion 2007, zwei Jahre Postdoc an der Harvard University in Boston. Als er im Jahr 2009 an die Technische Universität München auf eine Professur für Experimentelle Biophysik berufen wurde, war er gerade einmal 30 Jahre alt. Sogar sein Büro ist schmuck- und schnörkellos eingerichtet. Dunkle Ledersessel, schwarzes Metallregal, weiße Wände. Auf der Fensterbank stehen eine einsame Grünpflanze und das Modell eines Polyeders aus dem 3-D-Drucker. Weit und breit kein Foto, kein Buch und kein einziges Blatt Papier. Der Mann mit den adrett zurückgekämmten dunklen Haaren erinnert eher an einen Unternehmer als an einen Professor. Tatsächlich ist er längst beides: Gerade hat der 45-Jährige sein viertes Start-up gegründet.
Dietz gilt als weltweit führender Experte auf dem Gebiet des »DNA-Origami«. Selbst der Begründer der Technik, der US-amerikanische Chemiker Paul Rothemund, schreibt auf eine Interview-Anfrage hin, er selbst stehe derzeit nicht zur Verfügung, aber Hendrik Dietz sei »ein hervorragender Gesprächspartner zu dem Thema«. Rothemund hatte im Jahr 2006 demonstriert, dass es möglich ist, beliebig geformte Objekte aus DNA-Molekülen herzustellen. Dazu werden an einem langen DNA-Einzelstrang viele passgenaue kürzere Stränge befestigt, die diesen anschließend wie Heftklammern in die gewünschte Form falten. Die Videoanimation, die Dietz zur Veranschaulichung abspielt, zeigt, wie aus einer chaotischen Ansammlung von DNA-Sequenzen Ordnung wird - wie von Zauberhand.
Das Prinzip des DNA-Origami beruht darauf, dass sich die DNA-Basen Adenin, Cytosin, Guanin und Thymin immer zu komplementären Paaren zusammenfinden; und zwar stets in der energetisch günstigsten Konstellation. Adenin verbindet sich über Wasserstoffbrücken mit Thymin und Guanin mit Cytosin. So entsteht die typische DNA-Doppelhelix, deren Aussehen an eine Strickleiter erinnert. Es lässt sich entsprechend bereits am Computer mit geeigneter Software vorhersagen, welche Einzelstränge benötigt werden, um eine bestimmte Struktur zu bilden. »Dann stellt man alle DNA-Stränge her, kippt sie zusammen, erwärmt die Mischung, schüttelt – et voilà«, erklärt Hendrik Dietz. Wie ein Puzzle, das sich selbst zusammensetzt, entstehen durch programmierte Selbstorganisation ohne äußeren Eingriff die winzig kleinen Objekte. Unter dem Transmissionselektronenmikroskop lässt sich anschließend ihre Form sichtbar machen und kontrollieren, ob sich alles so gruppiert hat wie gewünscht.
Winzig kleine Bauklötze aus DNA
Was nach einer netten molekularbiologischen Spielerei klingt, elektrisiert die Fachwelt seit einigen Jahren. DNA-Moleküle, aus denen auch unsere Gene bestehen, sind kettenförmig, regelmäßig, stabil – und vor allem sehr gut erforscht. Seit James Watson und Francis Crick im Jahr 1953, also vor genau 70 Jahren, die Doppelhelix-Struktur der DNA aufgeklärt haben, experimentieren Scharen von Biologen damit. Mittlerweile lassen sich DNA-Sequenzen derart kostengünstig, schnell, hochrein und exakt herstellen, dass sie wie Legosteine als Baumaterial verwendet werden können. Allerdings sind die Bauklötze so winzig, dass man sie mit bloßem Auge nicht sehen kann. Nach Jahren der Grundlagenforschung scheinen nun endlich echte Anwendungen in greifbare Nähe zu rücken. Überall auf der Welt entstehen Start-ups, die versuchen, die Technik zu kommerzialisieren. Sie heißen »Capsitec«, »DoriVac«, »Twist Bioscience«, »Nanogami« oder »Nanovery«.
Der Erste, der erkannte, dass es möglich ist, Nanostrukturen aus DNA gezielt zusammenzusetzen, war der US-amerikanische Biophysiker Nadrian »Ned« Seeman. Der mittlerweile verstorbene Wissenschaftler gilt als der Begründer der DNA-Nanotechnologie. Er baute in den 1980er und 1990er Jahren aus synthetischen DNA-Strängen Nanoröhren, Polyeder, zwei- und dreidimensionale Gitter und andere Gebilde. Ursprünglich wollte er die Nanostrukturen als Gerüst für die Röntgenkristallografie verwenden, um beispielsweise Proteine, die allein nur schwer zu kristallisieren sind, in regelmäßigen Reihen anzuordnen. Doch die Technik, viele kurze Stränge in die gewünschte Form zu bringen, war aufwändig, fehleranfällig und schlecht reproduzierbar. Ihm fehlte die entscheidende Idee, die das Prinzip schließlich für unzählige, deutlich komplexere Anwendungen interessant machte: der lange Einzelstrang, auch Gerüst (englischen: scaffold) genannt, der von den kürzeren in Form gefaltet oder »getackert« wird.
Hendrik Dietz erinnert sich noch sehr gut an den Moment, als er das erste Mal von DNA-Nanotechnologie hörte. »Das war im Jahr 2003. Ich steckte gerade mitten in meiner Diplomarbeit, als ich einen Vortrag von Ned Seeman in München hörte«, schildert der Biophysiker. »Er stand da im tiefen Winter mit Sandalen und zeigte uns Bilder von Nanostrukturen wie etwa kleinen Würfeln, die aus einigen kurzen DNA-Stücken zusammengesetzt waren.« In seiner jugendlichen Arroganz habe ihn das nicht sonderlich beeindruckt. »Ich dachte, da ist doch nichts dabei. Das kann ich in 20 Sekunden nachbauen.« Damals habe er das für Spielerei gehalten.
Die Augen geöffnet habe ihm dann der Artikel im Fachmagazin »Nature« von Paul Rothemund im Jahr 2006. Die Forschungsarbeit schaffte es damals auf das Cover des Magazins. Abgebildet war ein grinsender Smiley aus DNA - in Wirklichkeit nur einige hundert Nanometer im Durchmesser. »Der Artikel war wegweisend«, sagt Hendrik Dietz. »Ich hatte gerade meine Doktorarbeit abgegeben, als ich den Harvard-Professor William Shih auf einer Konferenz in Venedig traf.« Man sei ins Gespräch gekommen. Schnell war klar, dass beide gleichermaßen von den neuen Möglichkeiten beeindruckt waren. »Rothemunds Idee war wirklich ein unglaublicher Technologiesprung«, sagt Dietz. Die Figuren waren allerdings zunächst noch flach und somit annähernd zweidimensional. Ihnen eine dritte Dimension und mehr Komplexität zu verleihen, habe ihn enorm gereizt, erklärt Dietz.
Er reichte einen Antrag für ein entsprechendes Forschungsprojekt bei der Deutschen Forschungsgemeinschaft ein – doch die lehnte zunächst ab. Die Begründung: Weil das Phosphat-Rückgrat der DNA negativ geladen ist, sei es auf Grund der elektrostatischen Abstoßung nicht möglich, auf diese Weise dreidimensionale Strukturen mit DNA-Origami zu bauen. »Rückblickend finde ich das sehr lustig«, sagt Dietz. Zusammen mit den beiden US-amerikanischen Wissenschaftlern William Shih und Shawn M. Douglas gelang es ihm dann während seiner Zeit an der Harvard University im Jahr 2009 zu zeigen, dass es sehr wohl möglich ist, dreidimensionale Strukturen mit DNA-Origami zu bauen. Um die Abstoßungskräfte zu überwinden, fügten die Forscher der Reaktionsmischung einen Überschuss an positiv geladenen Ionen hinzu, die die negativen Ladungen der DNA ausgleichen. Das habe »eine neue Ära auf dem Gebiet der strukturellen DNA-Nanotechnologie« eingeläutet, schrieb der Biochemiker Thomas LaBean von der Duke University in North Carolina damals in »Nature«. Die Wissenschaftler Yan Liu und Hao Yan von der Arizona State University äußerten sich ebenso begeistert in »Science«: »Es ist, als ob die DNA Yoga-Übungen erlernt hätte, um eine Vielzahl unterschiedlicher Haltungen im Nanomaßstab einzunehmen.« Dieser Erfolg half Hendrik Dietz dabei, eine Professur an der Technischen Universität München zu ergattern, wo er sich seitdem sukzessive seine Forschungsgruppe aufgebaut hat.
Der Traum von der Nanofabrik
Doch Dietz ist kein typischer Grundlagenforscher. »Mich befriedigt es nicht, im Labor etwas zusammenzurühren, das nichts taugt«, sagt er. »Mich interessiert immer die Frage: Was kann man damit machen?« Statt also Smileys oder geometrische Formen im Reagenzglas herzustellen, möchte er lieber kleine Maschinen im Nanometermaßstab bauen, die bestimmte Funktionen ausüben oder sogar völlig neue Stoffe wie beispielsweise maßgeschneiderte Medikamente herstellen können. In lebenden Zellen gibt es das zuhauf: etwa das Motorprotein Kinesin, das am Transport von biologischen Frachten innerhalb der Zellen beteiligt ist, oder Enzyme wie die ATP-Synthase, die Adenosintriphosphat (ATP), den Treibstoff der Zelle, herstellt. »Mein Traum ist es, eines Tages winzige Nanofabriken zu erstellen.«
Und die Verwirklichung dieses Traums rückt näher. Vor Kurzem beschrieb Dietz' Arbeitsgruppe in »Nature« einen DNA-Nanomotor, der tatsächlich messbare, mechanische Arbeit leisten kann. Die winzige Maschine funktioniert über einen Ratschenmechanismus, ähnlich wie Zahnräder in Uhrwerken. Wird eine Spannung angelegt, ist die Drehung in die eine Richtung energetisch günstiger als in die andere. Mikroskopische Aufnahmen zeigen, dass sich unter diesen Bedingungen jeder DNA-Arm des Motors – obwohl er sich zufällig schüttelt – im Schnitt immer in dieselbe Richtung dreht. Das sei ein wichtiger erster Beweis dafür, dass sich mit Hilfe der DNA-Origami-Technik nicht nur statische Nanosysteme herstellen lassen, sagt Hendrik Dietz.
Mit solchen Bestrebungen ist er nicht der Einzige. Überall auf der Welt gibt es mittlerweile Arbeitsgruppen, die DNA-Origami als etabliertes Werkzeug betrachten und nun nach geeigneten Anwendungen suchen. Darunter ist auch das Team von Kerstin Göpfrich. Die 32-jährige Biophysikerin leitet seit 2019 eine Forschungsgruppe am Max-Planck-Institut für medizinische Forschung und ist Professorin an der Universität Heidelberg. Die altehrwürdige Institution am Heidelberger Neckarufer hat fünf Nobelpreisträger hervorgebracht. Göpfrich will weg von den statischen DNA-Formen hin zu dynamischen Strukturen. »Jeder Student, der pipettieren kann, kann heute die DNA-Origami-Technik erlernen«, sagt sie. »Jetzt geht es darum, was man damit Sinnvolles tun kann.« Ihr Ziel ist es, eines Tages künstliche Zellen zu bauen. Doch anstatt nur das Leben, wie wir es kennen, zu kopieren, versucht sie, Zellen mit völlig neuen Bausteinen aus DNA-Origami zu entwickeln, die auf eine andere Weise als bislang funktionieren, Informationen weitergeben oder sich replizieren.
Der Forschungsbereich, in dem sie sich bewegt, nennt sich synthetische Biologie. Im so genannten Bottom-up-Ansatz wollen die Wissenschaftler, ausgehend von einfachen chemischen Bausteinen, komplexe Biosysteme herstellen. Dazu erforschen sie zunächst das Zusammenspiel einzelner Bestandteile einer Zelle: angefangen bei Zuckern und Fetten über Membranen bis hin zu Proteinen und Nukleinsäuren. Als Leitmotiv der Synthetischen Biologie gilt ein Zitat des Physik-Nobelpreisträgers Richard Feynman: »Was ich nicht herstellen kann, habe ich nicht verstanden« (im Original: »What I cannot create, I do not understand«).
Kerstin Göpfrich erklärt das Prinzip am Beispiel eines Toasters. »Wer einen Toaster nachbauen will, kann ihn auseinandernehmen, die einzelnen Teile nachahmen und dann alles wieder zusammensetzen. Das funktioniert, bietet aber keinen Vorteil. Was aber, wenn ich nur nach der wichtigsten Funktion schaue, nämlich ein Stück Brot zu rösten, und dann überlege, ob ich dieses Ziel nicht auch effizienter, besser und mit weniger Bauteilen erreichen kann? So gehen wir vor.« DNA-Origami sei dabei eine große Hilfe, etwa, um die einzelnen Module herzustellen, die für eine Zelle benötigt werden. Bis es tatsächlich so weit ist, wird es aber wohl noch eine Weile dauern.
DNA hat als Biomolekül jedoch eine weitere Eigenschaft, die sie vor allem für medizinische Anwendungen interessant macht: Sie bindet andere Biomoleküle. »DNA-Origami ermöglicht es uns, zum Beispiel Antigene in einer klar definierten Geometrie anzuordnen«, erklärt Kerstin Göpfrich. In einem Forschungsprojekt zusammen mit dem Deutschen Krebsforschungszentrum ahmen die Wissenschaftler die Verteilung und die Anzahl von Antigenen auf Virusoberflächen mit Hilfe von DNA-Origami nach, um herauszufinden, welchen Einfluss diese Parameter auf die Aktivierung der Antikörperproduktion haben. »Wir schauen, ob man Immunantworten verbessern kann, um so einen Tages bessere Impfstoffe etwa gegen Malaria herzustellen.«
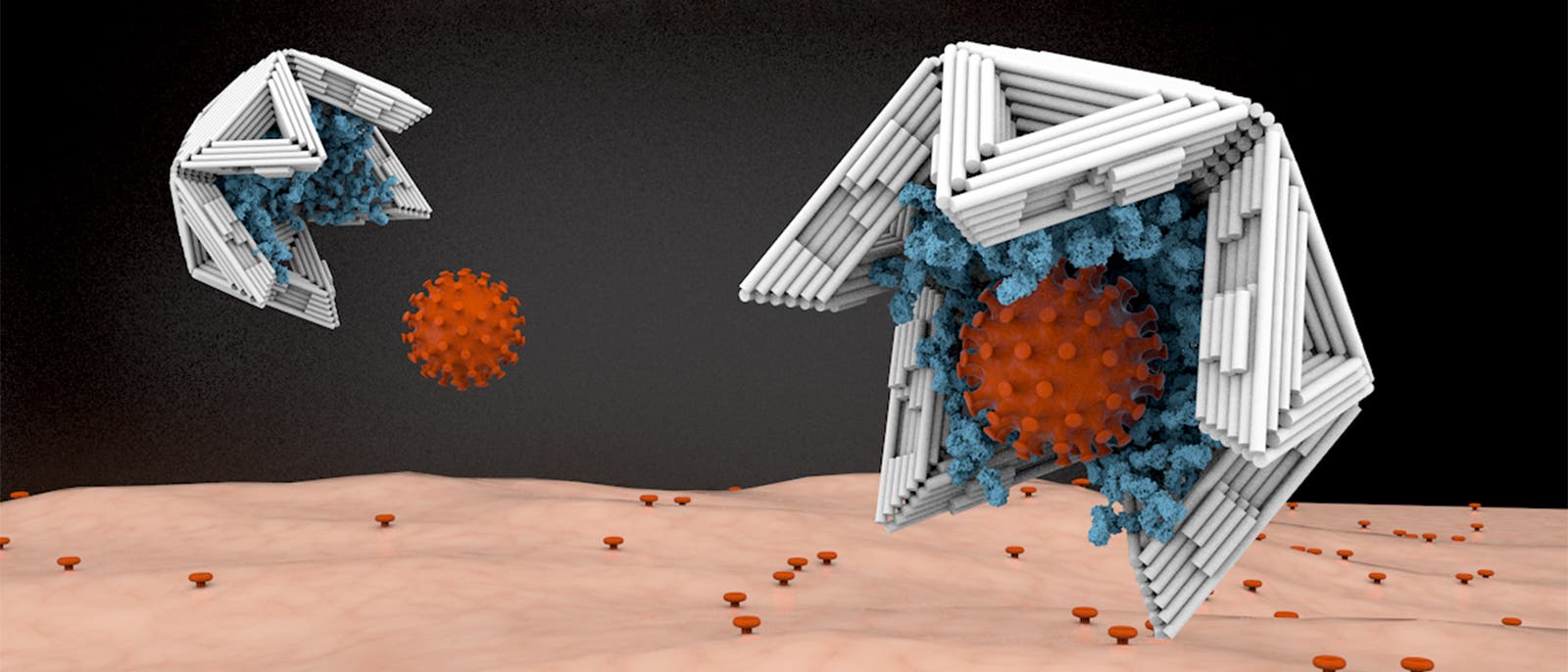
Viren in Quarantäne
Während sich dieses Projekt noch in der Frühphase befindet, hat das Münchner Start-up Capsitec bereits eine wichtige erste Hürde auf dem Weg zur kommerziellen Anwendung genommen: In-vivo-Studien im Mausmodell. Das Team um den Biophysiker Christian Sigl entwickelt derzeit ein Breitband-Virostatikum auf der Basis von DNA-Origami. Der 31-Jährige hat im Jahr 2022 seine Promotion in der Arbeitsgruppe von Hendrik Dietz abgeschlossen und anschließend gemeinsam mit Dietz und dem Virologen Ralf Wagner das Unternehmen gegründet, um die Technologie in die Anwendung zu bringen. Noch befindet sich Capsitec jedoch im Aufbau, selbst die Internetseite enthielt bis vor Kurzem noch Blindtext.
Das Prinzip, das ihrem Geschäftsmodell zu Grunde liegt, beschreiben sie im Fachmagazin »Nature Materials«. »Statt infizierte Menschen in Quarantäne zu schicken, nehmen wir die Viren gefangen und hindern sie daran, weitere Zellen zu infizieren«, erklärt Sigl. Dazu haben die Wissenschaftler ein programmierbares Baukastensystem aus baugleichen Dreiecken entworfen, die sich zu beliebig großen Schalen mit einem Innendurchmesser von aktuell bis zu 280 Nanometern zusammenstecken lassen - je nach Größe des Zielvirus. Auf der Innenseite sind die Käfige mit etlichen hundert virusspezifischen molekularen Bindungsstellen übersät. So sollen auch Krankheitserreger eingefangen werden können, für die nur Rezeptoren mit geringer Bindungsaffinität verfügbar sind. »Die Viren werden wie Fliegen in klebrigen Fliegenfallen unwiderruflich gefangen genommen, können sich dadurch nicht mehr vermehren und werden schließlich von der zelleigenen Müllabfuhr entsorgt«, sagt Sigl. Wissenschaftliche Veröffentlichungen oder begutachtete Ergebnisse aus den Mausversuchen gibt es bislang jedoch keine. Bis die Virenfallen im Menschen getestet werden dürfen, ist es daher noch ein weiter Weg.
»Die Viren werden wie Fliegen in klebrigen Fliegenfallen unwiderruflich gefangen genommen, können sich dadurch nicht mehr vermehren und werden schließlich von der zelleigenen Müllabfuhr entsorgt«Christian Sigl, Start-up-Gründer
Die Corona-Pandemie hat allerdings gezeigt, dass es manchmal auch ganz schnell gehen kann. Entsprechend versucht das Team von Capsitec, sich bereits jetzt auf dem Markt zu positionieren und Investoren von den Vorteilen der Methode zu überzeugen. Die im Auftrag des Bundesforschungs- und des Bundeswirtschaftsministeriums arbeitende Bundesagentur für Sprunginnovationen (SPRIN-D) unterstützt die Entwicklung im Rahmen des Wettbewerbs »Broad-spectrum Antivirals« mit insgesamt gut zwei Millionen Euro.
Der Vorteil eines solchen Medikaments etwa im Vergleich zu Impfungen sei, dass man viel schneller auf neue Pandemien und unbekannte Viren reagieren könne – unabhängig davon, wie viel man bereits über den Erreger weiß und ob er bereits mutiert, erklärt Sigl. Die kleinen Käfige können dem Konzept nach gleichzeitig Prophylaxe und Behandlung sein. Der Körper werde nicht wie bei einem Vakzin dazu angeregt, selbst Abwehrstoffe zu produzieren, betont Sigl. Stattdessen neutralisieren die DNA-Fallen die Viren bereits, bevor diese überhaupt in die Zellen eindringen können. »Alles läuft außerhalb der Zellen ab«, sagt er. »Das Einzige, was wir wissen müssen, sind die ungefähren Ausmaße des Virus.« Darauf könne dann die Größe der Käfige abgestimmt werden.
Für Hendrik Dietz ist Capsitec das vierte Start-up, das er gegründet hat - aber es scheint dasjenige zu sein, in das er die größten Hoffnungen steckt. »Es ist das wahrscheinlich komplizierteste und anspruchsvollste Medikament, das je entwickelt wurde«, sagt er. »Aber wenn es im Menschen wirklich funktioniert, dann wäre es ein echter Durchbruch.« Es wäre sozusagen das Meisterstück der DNA-Origami-Kunst.
Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.