Lexikon der Biologie: Nitrogenase
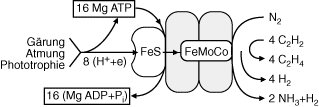
Nitrogenasew [von Nitrogenium], Multienzymkomplex, der die Umwandlung von molekularem Stickstoff (N2) zu Ammoniak (NH3), d.h. die Stickstoffixierung, katalysiert ( vgl. Abb. 1 ). Der Nitrogenase-Komplex besteht aus 2 Funktionseinheiten, der Dinitrogenase-Reductase (ursprünglich auch Azoferredoxin genannt), welche die Dinitrogenase (ursprünglich auch Molybdoferredoxin genannt), an der die Reduktion von molekularem Stickstoff (Dinitrogen = N2) stattfindet, mit den erforderlichen Reduktionsäquivalenten (Redoxreaktionen) versorgt. Bislang sind 3 verschiedene Nitrogenase-Komplexe untersucht worden: der sog. konventionelle und 2 alternative Komplexe. Bei allen 3 Formen besteht die Dinitrogenase-Reductase aus einem weitgehend gleich aufgebauten Eisen-Schwefel-Protein. Die Dinitrogenase der konventionellen Molybdän-Nitrogenase besteht dagegen aus je 1 Paar von 2 verschiedenen Proteinen (α2β2) und dem Eisen-Molybdän-Cofaktor als prosthetischer Gruppe (Molybdän, Eisen, Schwefel). Rhizobien, besser als Knöllchenbakterien bekannt, vermögen nur die konventionelle Nitrogenase zu synthetisieren. Im Gegensatz dazu wird bei Molybdän-Mangel von einigen Bakterien (Azotobacter vinelandii, Azomonas agilis, Azotobacter chroococcum; Azotobacter, Azomonas) alternativ die Vanadium-Nitrogenase gebildet, die aus einem Hexamer aus 3 verschiedenen Proteinen (α2β2δ2) mit dem Eisen-Vanadium-Cofaktor besteht. Fehlen sowohl Molybdän als auch Vanadium, so sind wiederum einige Bakterienstämme (Azotobacter vinelandii, Azomonas agilis) in der Lage, die Nur-Eisen-Nitrogenase mit dem Eisen-Eisen-Cofaktor eines hexameren Apoproteins aus 3 Proteinen, zu synthetisieren. Es gibt auch Bakterien (Rhodobacter capsulatus, Rhodospirillum rubrum; schwefelfreie Purpurbakterien), die bei Molybdän-Mangel ausschließlich den Nur-Eisen-Nitrogenase-Komplex ausbilden. Die 3 Formen der Nitrogenase werden von 3 verschiedenen Gensätzen codiert, die folgende Bezeichnungen tragen: nif für die Gene der Molybdän-Nitrogenase, vnf für die Vanadium-Nitrogenase und anf für die Nur-Eisen-Nitrogenase (nif-Gene, nif-Regulon). Die Nitrogenase-Reaktion erfordert 8 Reduktionsäquivalente und Energie in Form von mindestens 2 ATP (Adenosintriphosphat) pro übertragenem Elektron ( vgl. Abb. 2 ). Die Reduktionsäquivalente und ATP werden von den Organismen je nach ihrer Zugehörigkeit zu verschiedenen Stoffwechseltypen bereitgestellt (Atmung, Chemotrophie, Gärung, Phototrophie). Zwischenstufen bei der Reduktion von N2 zu 2 NH3 sind wahrscheinlich das Diimid und Hydrazin, die jedoch beide im enzymgebundenen Zustand mit den verfügbaren Methoden nicht nachweisbar sind. Im Verlaufe dieser Reaktion wird pro umgesetztem N2 immer auch ein H2 (Wasserstoff) freigesetzt. Die gängige Erklärung hierfür ist, daß zunächst 2 Wasserstoff-Ionen von dem Enzym aufgenommen werden müssen, bevor N2, dies dann unter Freisetzung von H2, gebunden werden kann. Es gibt Organismen, die bei einem Mangel an N2 im Verlaufe eines Funktionszyklus der Nitrogenase sämtliche Elektronen auf Protonen übertragen und somit 4 H2 anstelle von üblicherweise 2 NH3 und 1 H2 entwickeln (photobiologische Wasserstoffbildung). – Nitrogenase kann neben N2 eine Reihe anderer Substrate mit Mehrfachbindungen reduzieren ( vgl. Abb. 3 ). Darunter ist die Reduktion von Acetylen (HC≡CH; Alkine) zu Ethylen (H2C=CH2) die geläufigste Methode für die Ermittlung der Nitrogenase-Aktivität. Im Gegensatz zur konventionellen Nitrogenase lassen sich die alternativen Nitrogenasen relativ einfach anhand ihrer Fähigkeit identifizieren, Acetylen zu Ethylen und Ethan zu reduzieren. Die Stickstoffixierung und die Ausbildung des hierfür notwendigen Funktionssystems stellen recht hohe Anforderungen an den Stoffhaushalt der Zellen. Es ist deshalb nicht verwunderlich, daß sowohl die Ausbildung der Nitrogenase als auch ihre Aktivität strikten Kontrollmechanismen unterworfen sind. Die bei weitem wichtigsten Faktoren für die Regulation der Nitrogenase sind die Gegenwart von gebundenem Stickstoff und von Sauerstoff. Während die Bedeutung von gebundenem Stickstoff für die Regulation ohne weiteres einleuchtend ist, erklärt sich die Regulation durch Sauerstoff damit, daß die Nitrogenase in Gegenwart von Sauerstoff innerhalb weniger Minuten irreversibel zerstört werden kann. Besonders solche Organismen, die in Gegenwart von Sauerstoff Stickstoff fixieren, haben deshalb Mechanismen zum Schutz der vorhandenen Nitrogenase entwickelt. Diese Mechanismen können je nach Organismus verschieden sein. Die Grundlage hierfür ist jedoch, daß die Nitrogenase bei einer plötzlichen Erhöhung der Sauerstoff-Konzentration (Sauerstoff-Streß; oxidativer Streß) in einen geschützten Zustand überführt, dabei aber in der Aktivität reversibel abgeschaltet wird. Wird der Streß aufgehoben, so setzt die Aktivität wieder ein. Hält der Streß dagegen an, so wird die Synthese der Nitrogenase unterbunden (nif-Regulon). Eisen-Nitrogenasen, Heterocysten, Leghämoglobin.
J.O.

Nitrogenase
Abb. 1: Reduktion von Stickstoff (N2) zu Ammoniak (NH3) durch Nitrogenase

Wenn Sie inhaltliche Anmerkungen zu diesem Artikel haben, können Sie die Redaktion per E-Mail informieren. Wir lesen Ihre Zuschrift, bitten jedoch um Verständnis, dass wir nicht jede beantworten können.